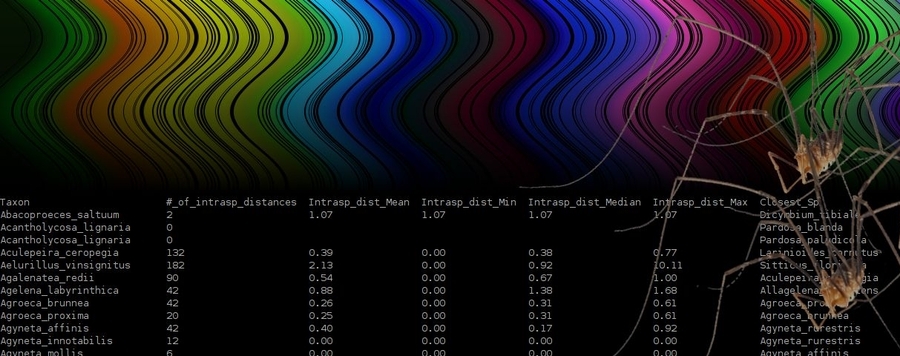
DiStats
Das Perl-Skript DiStats vereinfacht die Verarbeitung von DNA-Barcode-Daten und Erstellung von Daten-Releases. Neben anderen Statistiken berechnet es intraspezifische Distanzen, die am nächsten verwandten Artenpaare, oder die am weitesten entfernten Congener (Arten aus derselben Gattung). DiStats verarbeitet Sequenzen im FASTA-Format und berechnet p-Distanzen oder K2P-Distanzen. DiStats kann zwei verschiedene Ausgabeformate produzieren: a) eine Tabelle mit Statistiken für jede Art sowie b) eine Matrix mit allen paarweisen Distanzen im Datensatz. Für einen Alignment-Datensatz mit 1.000 COI Barcode-Sequenzen dauert die Analyse ca. 6 Minuten (single-threaded mit einem 3,4 GHz Prozessor). DiStats hat eine algorithmische Komplexität von O(n^2), was bedeutet, dass die Laufzeit exponentiell mit der Zahl der Sequenzen im Datensatz (n) ansteigt. DiStats kann parallelisiert werden: durch die Nutzung von multi-threading reduziert sich die Laufzeit um einen Faktor von 1/c (c = Anzahl an CPU-Threads).
Dokumentation im Download-Paket inbegriffen.
Zitation:
Astrin, Höfer, Spelda, Holstein, Bayer, Hendrich, Huber, Kielhorn, Krammer, Lemke, Monje, Morinière, Rulik, Petersen, Janssen, Muster (2016). Towards a DNA barcode reference database for spiders and harvestmen of Germany. PLoS ONE 11(9): e0162624. doi:10.1371/journal.pone.0162624
Programmierung:
Hannah Janssen & Malte Petersen






