COGNATE
COGNATE: Comparative Gene Annotation characterizer
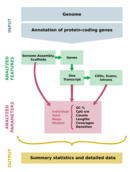
COGNATE ist ein Programm zur simultanen Analyse einer gegebenen Annotation von protein-kodierenden Genen und den dazugehörigen assemblierten Genomsequenzen. Es erlaubt die schnelle und einfache Extraktion basaler Genomeigenschafts- und Genrepertoiredaten. Es ist demnach ein Werkzeug für die Primärbeschreibung eines Genoms und seiner Genannotation; dies ist eine Grundvorraussetzung für vergleichende und übergreifende Analysen von Genom- und Genstruktur.
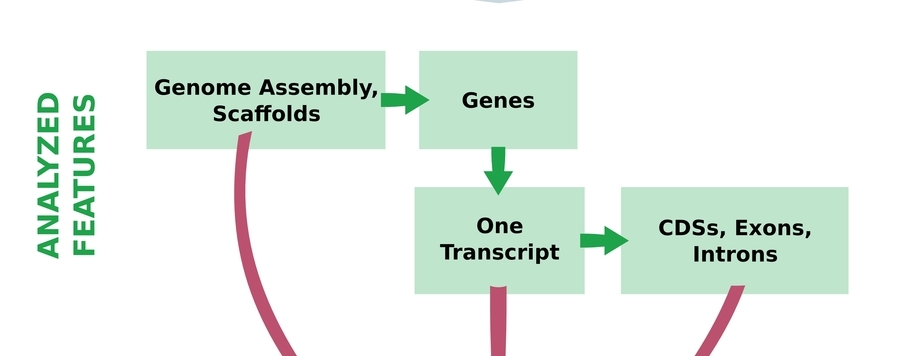
COGNATE erschließt die folgenden Hauptparameter:
- Anzahlen der analysierten Merkmale
- Leserichtung von Transkripten und deren CDSs, Exons und Introns
- Längen und Längenstatistiken (Nukleotid-/Aminosäuresequenzen), einschließlich N50/L50, 75/L75, N90/L90
- Intronlängenverteilung wie vorgeschlagen bei [1]
-
prozentualer GC-Gehalt, berechnet auf zwei Arten:
- mittels einer Formel, die explizit IUPAC Ambiguitätskodes berücksichtigt (G, C, S pro Gesamtlänge weniger N, R, Y, K, M, B, D, H, V)
- mittels der vorher vorherrschenden Formel, die GC pro Gesamtlänge berechnet; diese ist ungeeignet für Genomvergleiche, da das Ergebnis von der Assemblyqualität abhängt
- Statistiken der CpG-Dinukleotid-Abreicherung (CpG beobachtet/erwartet), normalisiert durch den Gehalt von G und C in der betrachteten Region [2]
- Dichte-Statistiken (Verhältnis der Bedeckung eines Merkmals durch ein anderes, anzahlmäßig)
- Bedeckungs-Statistiken (Verhältnis der Bedeckung eines Merkmals durch ein anderes, längenmäßig)
Das Ausgabedesign ist auf Klarheit und die Kombination von Übersichtsdarstellung und detaillierter Parameterauswertung ausgerichtet, um COGNATE so nützlich wie möglich innerhalb des ausgewiesenen Anwendungsbereiches zu machen.
Für die Benutzung von COGNATE werden eine funktionierende Installation von Perl sowie GAL [3] benötigt. GAL ist von Barry Moore programmert worden und wurde ursprünglich durch die Sequence Ontology [4] bereitgestellt. Der von COGNATE benötigte Ordner (GAL/lib/) ist, mit kleineren Änderungen, in dem COGNATE-Paket enthalten (als GAL/) und muss nicht weiter installiert/behandelt werden.
Für Fehlerberichte und Anfragen zu Erweiterungen oder alten Versionen kontaktieren Sie bitte Jeanne Wilbrandt oder Bernhard Misof.
Zitation
Wilbrandt J, Misof B, Niehuis O (2017) COGNATE: Comparative Gene Annotation characterizator. BMC Genomics 18:535.
https://github.com/ZFMK/COGNATE
Quellen
- Roy SW, Penny D. Intron length distributions and gene prediction. Nucl. Acids Res. 2007;35:4737–42.
- Elango N, Hunt BG, Goodisman MAD, Yi SV. DNA methylation is widespread and associated with differential gene expression in castes of the honeybee, Apis mellifera. PNAS. 2009;106:11206–11.
- Genome Annotation Library, https://github.com/The-Sequence-Ontology/GAL
- Eilbeck K, Lewis SE, Mungall CJ, Yandell M, Stein L, Durbin R, et al. The Sequence Ontology: a tool for the unification of genome annotations. Genome Biology. 2005;6:R44.
Abbildung
Übersicht der Arbeitsabläufe und analysierten Parameter in COGNATE.